
王彬1,王秀2,严斌斌1,翟英男1,马丽1,王国臻1,包云英3,李明成1
(1:北华大学医学技术学院, 吉林 132013;2:锦州医科大学临床医学院, 锦州 :121001
3:北华大学附属医院 吉林 132013)
摘要:目的:探讨肠癌患者与健康人群肠道中大肠埃希菌硝基还原酶(the Eschenichia coli major oxygen-insensitive nitroreductase ,NfsA)基因特征及酶活性差异。方法:采集31例肠癌患者及30例健康人群粪便标本,鉴定、分离出E.coli并设计特异性引物扩增NfsA基因,采用克隆测序技术比较肠癌患者和健康人群肠道NfsA基因序列差异;采用酶-特异性底物法比较肠癌患者和健康人群肠道NfsA活性差异。结果:31例肠癌患者及30例健康人群肠道粪便标本经分离、鉴定均为E.coli.肠癌患者肠道中NfsA基因序列相对于标准NfsA基因序列在第650个碱基处由G突变成A、在第41个碱基处由G突变成A、在第369个碱基处由A突变成T、在第72个碱基处由A突变成C,NfsA的活性高于健康人群对照组具有统计学差异(P<0.05)结论:肠癌患者肠道NfsA在基因序列及酶活性均与健康人群存在差异,这与结直肠癌的发生、发展密切相关,可能进一步导致了结直肠癌的发生、发展。
中图分类号:R735.3+7 文献标示码:A
作者简介:王彬(1994-),男,河北沧州人,硕士研究生在读,研究方向:病原微生物基因检测 E-mail:1505646848@qq.com
通讯作者:李明成(教授),E-mail:576466156@qq.com
基金项目:吉林省卫生和计划生育委员会资助项目(2018j098)
关键词:培养、鉴定E.coil; NfsA基因序列差异 ; NfsA活性差异.
Identification and characterization of nitroreductase gene of Escherichia coli in intestinal tract of patients with colorectal cancer and healthy persons
Abstract:To investigate the difference of NfsA gene, enzyme activity in intestinal tract between patients with colorectal cancer and healthy subjects.Methods:Stool samples from patients with colorectal cancer and healthy people were collected, and Escherichia coli was selectively cultured, identified and isolated.Specific primers were designed and NfsA genes were amplified. Cloning and sequencing techniques were used to analyze the gene sequence differences between colorectal cancer patients and healthy controls.The difference of enzyme activity was analyzed by enzyme-specific substrate method.Results:Intestinal stool samples were selectively cultured and identified as Escherichia coli. Compared with standard NfsA gene sequence, NfsA gene sequence of intestinal cancer patients was mutated from G to A at 650th base, from G to A at 41st base, from A to T at 369th base. The activity of NfsA in fecal samples of patients with colorectal cancer was significantly higher than that in control group (P < 0 05).Conclusion:The mutation, expression and activity of NfsA gene in feces of patients with colorectal cancer are closely related to the occurrence and development of colorectal cancer, and further lead to the occurrence and development of colorectal cancer.
Key words:Culture and identification of Escherichia coli; NfsA sequence difference; NfsA activity difference
近年来,人们患恶性肿瘤的概率持续增加【1】。大肠癌是最为常见的恶性肿瘤之一,是全球第四大癌症,尽管在预防及治疗方面取得了进展,但仍然是继肺癌之后第二大致命肿瘤,并且结直肠癌的发病率在青少年和年轻成人(AYA)患者中持续增加,这些患者被定义为45岁以下的个体【2】【3】。其中饮食习惯、家族遗传、生活环境是CRC发生的重要条件【4】【5】。肠道菌群与肠癌的发生、发展是当今研究的热点。
最近的许多工作都集中在胃肠道微生物群上,这是一种复杂的共生菌群,由1014个单独的细菌和大于300万个基因组成【6】。肠道微生物群,深刻的影响宿主生理学稳态,包括营养代谢,抗感染和免疫系统发育等方面【7】。肠道微生物编码合成的硝基还原酶存在与多种细菌中且含量丰富,以肠杆细菌为主。主要有大肠埃希菌(Eschericha.coli)【8】、哈维氏弧菌(Vibrio harveyi)【9】、恶臭假单胞菌(Pseudomonas putida)【10】、沙门氏菌(salmonella)【11】、阴沟肠杆菌(Enterobacter cloacae)【12】等。其中E.coil是人类肠道微生物中的一种常见细菌,在维持肠道稳态方面起着重要作用【13】。大肠埃希菌体内的硝基还原酶(NfsA)可以将人类胃肠道中硝酸盐还原为具有活性和毒性的亚硝酸盐,亚硝酸盐进一步与含氮化合物结合后产生亚硝基化合物,亚硝基化合物是高度致癌物。同时研究表明,N-亚硝基化合物与人类基因表达改变有关,证明了这些化合物在大肠癌发生、发展中的潜在作用【14】【15】。本文就肠癌患者及健康人群肠道E.coil携带的NfsA在基因序列及酶活力方面做一研究。
1.研究对象和方法
1.1研究对象 收集自2018年5月至2019年9月到吉林市肿瘤医院就诊的未进行手术及放化疗治疗之前大肠癌患者粪便标本31例。其中男患者19例,女患者12例。年龄在43-77之间,平均年龄是(61.33±9.88)。同时收集同期医院体检中心健康人群粪便标本30份。其中男17例,女13例,年龄在45-67之间,平均年龄为(57.58±5.79)。肠癌患者及健康对照者,在性别分布及年龄构成等一般资料方面均无显著差异(P>0.05)
本研究中结直肠癌患者的统计和排除标准为:①患者的临床症状符合结直肠癌诊断标准,并且经过病理学及血清学检查确诊为大肠癌。②无消化道疾病(如胰腺炎、肠梗阻等)④未经过结直肠癌手术治疗和放化疗等辅助治疗。③对本研究知情并且同意的患者。⑤排除机体功能有严重损害者(如心、肺、肝等器官功能损害)⑥排除家族遗传性消化道疾病(如大肠腺瘤、肠道炎症性疾病等)⑦排除近一月肠道菌群可能分布及代谢紊乱的患者(如服用抗生素、微生态制剂治疗等)。健康对照者统计和排除的标准为:①无家族性或慢性肠道疾病的健康对照着。②体检各项指标均在正常范围内的健康对照者。③近期内肠道菌群分布及代谢正常者。④对本研究知情并且同意者。
1.2实验方法
1.2.1细菌的分离培养与鉴定 ①收集肠癌患者及健康对照者新鲜粪便并在0.5h内送检,按照E.coil检测标准流程鉴定,最后进行16SrDNA进行基因测序鉴定。
1.2.2引物的合成与PCR扩增 由上海生工设计并合成16SrDNA引物、扩增NfsA基因引物,并在NCBI上检测其特异性,然后进行梯度PCR,探索最适退火温度。用细菌基因提取试剂盒提取大肠杆菌基因组。然后进行PCR(反应体系25μl:2×Taq Mix 12.5μl 上游引物 0.5μl 下游引物 0.5μl 目的基因 1μl. 循环参数:预变性95℃ 5min、变性95℃ 30S 退火55℃ 30s、延伸72℃ 30s 30个循环 充分延伸72℃ 5min)扩增出NfsA基因全长并进行电泳,用琼脂糖凝胶纯化试剂盒将电泳后的DNA片段提纯.
1.2.3克隆测序技术检测肠癌患者NfsA基因序列 用pGM-T克隆试剂盒将NfsA基因连接到载体上,加入到TOP10感受态细胞中,并向管中加入500μl预热的SOC培养液,37°培养3h使菌体复苏。吸取100μl混匀的菌液接种于转化平板上,过夜培养。将平板上白色菌落接种到6ml的LB(含有80μg/ml的Amp)液体培养基,37℃、150r/min震荡过夜培养。用质粒提取试剂盒提取菌液中的质粒,经PCR扩增和电泳验证成功后,送到上海生物工程有限公司进行测序,并在NCBI上与标准NfsA基因序列进行比对。
1.2.4酶-底物法检测NfsA活性 ①将NfsA的特异性底物(呋喃西林粉)溶于PBS缓冲液配制成0.1mM,并梯度稀释成0.1-0.01mM,用酶标仪在25℃,波长410nm处测得吸光值。根据吸光度及其浓度制作标注曲线。②挑取单个菌落接种于发酵培养基中150r/min,10h震荡过夜培养,吸取200ml与96孔板中在600nm波长处测得吸光度为0.7.然后4000r/min、2min离心。将NfsA的特异性底物与诱导剂ITTG溶解于PBS缓冲中配制成浓度分别为0.1mM、0.2mM的溶液。吸取50μl离心后的菌液和150μl含有特异性底物与诱导剂的PBS缓冲液加入到200μl的96孔板中,吹打混匀放入到37℃培养箱中,培养5h。③然后用酶标仪25℃、410nm波长处测得吸光值,根据标准曲线计算出浓度。根据酶活力定义,按照公式U/L=(C始-C末)×V/h计算出肠癌患者和健康对照者NfsA活力。
1.2.5 统计结果
采用SPSS17.0软件进行统计学处理、分析。实验数据采用`X±S表示,组内t检验,P<0.05表示具有统计学差异。
2结果
2.1 细菌鉴定结果 61份粪便标本细菌经形态学及生化反应及16sRDNA测序鉴定,符合大肠癌埃希菌的特征。与Gene Bank收录中的Escherichia fergusonii(弗氏大肠杆菌) ATCC 35469菌株相符(图1)。

图1 粪便标本可疑菌落16sRDNA测序结果
2.2 肠癌患者及健康人群肠道NfsA基因扩增结果
2.2.1 肠癌患者粪便标本NfsA基因扩增结果:肠癌患者及健康对照者粪便标本大肠杆菌基因组经PCR扩增、电泳后均在723bp处出现了明亮的目的基因条带(图2)。
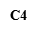



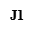

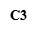
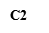



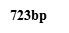








M:D2000Marker;Y:阴性对照;C1-C7:肠癌患者粪便标本;J1健康对照者粪便标本
图2 肠癌患者及健康人群肠道E.coil携带的NfsA的琼脂糖凝胶电泳图谱
2.3肠癌患者及健康人群肠道E.coil携带的 NfsA基因测序及对比结果.
重组质粒经上海生物工程有限公司测序并在NCBI上和标准的NfsA基因对比后发现肠癌患者肠道E.coil携带的NfsA基因第650个碱基处由G突变成A、在第41个碱基处由G突变成A、在第369个碱基处由A突变成T,部分测序结果(图3)。


图3 部分肠癌患者肠道NfsA测序比对结果
2.4 肠癌患者及健康人群肠道E.coil携带的NfsA活性结果及分析
2.4.1 酶-底物法绘制呋喃西林粉标准曲线结果:Y=1.9866x+0.0493。相关系数r2=0.9996,相关性良好,(图4)。

图4呋喃西林粉标准曲线
2.4.2 统计学分析结果:
与健康对照组相比,CRC组NfsA活性明显升高,并且具有统计学差异(P<0.05),表1。
表1 NfsA活性(`X+±s,U/L)
|
分组 |
NfsA活性 |
|
CRC |
46.73±5.02 |
|
健康对照组 |
40.23±4.46 |
|
F |
0.084 |
|
P |
<0.05 |
注:健康对照组与CRC组比较*P<0.05.
3讨论:
结直肠癌症是一种多因素疾病,涉及遗传和表观遗传改变、环境因素和生活方式等方面【16】。同时癌症基因学研究为观察癌症发展中的错综复杂性提供了一种可靠的视角【17】【18】。越来越多的研究说明肠道微生物的改变与结直肠癌的发展密切相关【19】。Mira-Pascual【20】 等研究发现结直肠癌患者与健康人群菌群结构不同,结直肠癌组织中存在较多的核酸杆菌和肠杆菌。Bullman 等[21]研究显示梭杆菌属还与肿瘤的发展、恶化、转移有关。较多研究同时也有研究表明细菌的代谢产物次级胆汁酸如脱氧胆酸能产生活性氧自由基可导致肠粘膜细胞的DNA断裂及染色体不稳定进而诱导肿瘤的发生【22】【23】。丁酸是一种短链脂肪酸,可以增强免疫、减轻炎症、阻止DNA甲基化,保护DNA不受损伤,因此有阻止结直肠癌的作用【24】。Tirandaz等【25】研究发现能够产生丁酸的细菌如所装芽孢杆菌和罗氏军属在结直肠肿瘤中表达量低于健康组。由于肠道微生物与结直肠癌的特殊关系,肠道微生物及其代谢产物在癌症的检测及治疗具有重要作用【26】。
其中进行饮食干预是最为常见的方法之一具有很高的可行性和安全性,通过饮食来控制肠癌的发病率及改善患癌后的恶化情况【27】。例如,美国研究学者发现长期大量摄入鱼类的人群相对于正常人群患CRC的风险率明显降低,这可能是由于鱼中含有ω-3和ω-6多元不饱和脂肪酸(PUFA)具有抗炎作用有关【28】。摄入膳食及谷类等植物性食物可预防CRC的发生率,这一机制可能与膳食纤维经细菌发酵产生脂肪酸等抗癌物质有关【29】【30】。同时研究学者还发现高脂饮食导致小鼠表达过量的脂肪因子和细胞因子增加了患CRC的几率,以及红肉摄入量过多进而在肠道内产生致癌物质也会增加正常人群患CRC的风险【31】【32】【33】。另一方面,肠道菌群也可以在传统的放化疗方面发挥作用。Iida【34】等通过小鼠模型证实,肠道菌群缺陷的荷瘤小鼠对奥沙利铂药物治疗无效果,而对肠道菌群健康的小鼠有治疗效果。同时研究学者还发现,顺铂和抗生素联合用药加快了小鼠肿瘤组织的生长,减低了小鼠的存活时间。而顺铂和乳酸菌联合用药时,会抑制肿瘤细胞的增长【35】。而给患者补充益生菌和粪便微生物群移植(FMT)也是热门的研究之一。如丁酸杆菌和枯草芽孢杆能够产生丁醇的细菌,在小鼠模型中可抑制了DMH(二甲基肼二盐酸盐)诱导的CRC的发展,该分子机制涉及降低炎症反应和保持免疫稳态【36】。另一种益生菌干酪乳杆菌(菌株BL23)不仅在小鼠模型中成功的抑制了CRC的发展,而且调节肠道菌群向健康的稳态发展【37】。另外个体间肠道菌群的交换已被用于治疗病原体感染或肠道炎症性疾病和生物失调。Khoruts【38】等通过对272位感染艰难梭菌临床患者的实验分析证明FMT用于治疗复发性艰难梭菌十二指肠感染是有效的。而最近一项临床实验证明,5例临床患者使用阴道菌群移植(VMT)治疗难治性细菌阴道炎,其中4例完全缓解,1例部分缓解,且在5名临床患者中没有观察到任何不良反应【39】。因此,随着我们对微生物治疗学有了更深入的了解,未来的发展很可能会从全粪便移植到更精细和更加特异性的细菌移植,已达到临床治疗效果【40】。
所有研究提示结直肠癌的发生、发展与肠道微生物的种类、数量有关及代谢产物有关。因此,如何通过监测肠道微生物及其代谢产物的变化来预测结直肠的发生发展,从而达到早期预防、早期治疗的效果这一问题迫在眉睫。本研究发现肠癌患者肠道E.coil携带的NfsA在基因序列活性方面,相对于健康人群均发生了改变。为肠道菌群与结直肠的研究提供了一种新视角,为肠癌的早期诊断提供了一种无创性指标。但是,本研究采取的肠癌患者样本量较少,且没有对肠癌患者病情进行明显分期。因此,今后应加大样本量,并且对肠癌患所患恶性肿进行TNM分期,进一步完善本研究。
1.Brody H. Colorectal cancer. Nature[J].2015 14;521(7551)
2.Rebecca S, Carol D, Ahmedin Colorectal cancer statistics, 2014[J]. Ca A Cancer Journal for Clinicians, 2014, 64(2):104-117.
3.Weinberg B A , Marshall J L , Salem M E . The Growing Challenge of Young Adults With Colorectal Cancer[J]. Oncology (Williston Park, N.Y.), 2017, 31(5):381-389.
4.Jobin, C. Colorectal Cancer: Looking for Answers in the Microbiota[J]. Cancer Discovery, 2013, 3(4):384-387.
5.Jobin C. Human Intestinal Microbiota and Colorectal Cancer: Moving Beyond Associative Studies[J]. Gastroenterology. 2017 ,153(6):1475-1478.
6.Abreu M T , Peek R M . Gastrointestinal Malignancy and the Microbiome[J]. Gastroenterology, 2014, 146(6):1534-1546.e3.
7.Fung T C , Olson C A , Hsiao E Y . Interactions between the microbiota, immune and nervous systems in health and disease[J]. Nature Neuroscience, 2017, 20(2):145-155.
8.Yang J , Zhan J , Bai J , et al. Residue Phe42 is critical for the catalytic activity ofEscherichia colimajor nitroreductase NfsA[J]. Biotechnology Letters, 2013, 35(10):1693-1700.
9.Kwak Y H , Lee D S , Kim H B . Vibrio harveyi Nitroreductase Is Also a Chromate Reductase[J]. Applied and Environmental Microbiology, 2003, 69(8):4390-4395.
10.Antonio C , Juan J, Lázaro, Ramos J L , et al. PnrA, a new nitroreductase-family enzyme in the TNT-degrading strain Pseudomonas putida JLR11[J]. Environmental Microbiology, 2010, 7(8):1211-1219.
11.Yanto Y , Hall, Mélanie, Bommarius A S . Nitroreductase from Salmonella typhimurium: characterization and catalytic activity[J]. Organic & Biomolecular Chemistry, 2010, 8(8):1826-32.
12.Isayev O , Carlos E. Crespo‐Hernández, Gorb L , et al. In silicostructure–function analysis of E. cloacae nitroreductase[J]. Proteins: Structure, Function, and Bioinformatics, 2012, 80(12):2728-2741.
13. Leser TD, Mølbak L. Better living through microbial action: the benefits of the mammalian gastrointestinal microbiota on the host[J]. Environ Microbiol. 2009 ,11(9):2194-206.
14:Hebels DG,Sveje KM,de Kok MC,et al. N-nitroso compound expo- sure-associated transcriptomic profiles are indicative of an increased risk for colorectal cancer [J]. Cancer Lett,2011,309( 1) : 1-10.
15.Kuugbee E D , Shang X , Gamallat Y , et al. Structural Change in Microbiota by a Probiotic Cocktail Enhances the Gut Barrier and Reduces Cancer via TLR2 Signaling in a Rat Model of Colon Cancer[J]. Digestive Diseases and Sciences, 2016, 61(10):2908-2920.
16. Pope JL, Tomkovich S, Yang Y,et.al. Microbiota as a mediator of cancer progression and therapy.[J].Translational Research. 2017 ,179:139-154.
17.Vogelstein B, Papadopoulos N, Velculescu V E, et al. Cancer Genome Landscapes[J]. Science, 2013, 339(6127):1546-1558.
18.Wheeler D A , Wang L . From human genome to cancer genome: The first decade[J]. Genome Research, 2013, 23(7):1054-1062.
19.Tahara T , Arisawa T . Pathogenesis of CpG island methylator phenotype-positive colorectal cancers: role of genetic alteration and colonic flora[J]. Epigenomics, 2014, 6(3):249-251.
20.Mira-Pascual L , Cabrera-Rubio R , Ocon S , et al. Microbial mucosal colonic shifts associated with the development of colorectal cancer reveal the presence of different bacterial and archaeal biomarkers[J]. Journal of Gastroenterology, 2015, 50(2):167-179.
21.Bullman S , Pedamallu C S , Sicinska E , et al. Analysis of Fusobacterium persistence and antibiotic response in colorectal cancer[J]. Science. 2017,358(6369):1443-1448.
22.Setyawati M . Azcárate-Peril MA, Sikes M,et.al. The intestinal microbiota, gastrointestinal environment and colorectal cancer: a putative role for probiotics in prevention of colorectal cancer[J]. Am J Physiol Gastrointest Liver Physiol Am J Physiol Gastrointest Liver Physiol, 2011 ,301(3):G401-24.
23.Elinav E , Nowarski R , Thaiss C A , et al. Inflammation-induced cancer: crosstalk between tumours, immune cells and microorganisms[J]. Nature Reviews Cancer, 2013, 13(11):759-771.
24.Worthley D L , Whitehall V L J , Leu R K L , et al. DNA Methylation in the Rectal Mucosa Is Associated with Crypt Proliferation and Fecal Short-Chain Fatty Acids[J]. Digestive Diseases & Sciences, 2011, 56(2):387-396.
25.Tirandaz H , Mohammadi E . Efficient tumor targeting by anaerobic butyrate-producing bacteria[J]. Medical Hypotheses, 2013, 80(5):675-678.
26.McQuade JL, Daniel CR, Helmink BA, et.al. Modulating the microbiome to improve therapeutic response in cancer[J]. Lancet Oncol. 2019 ,20(2):e77-e91.
27.Blottière, Hervé M, De Vos W M , Ehrlich S D , et al. Human intestinal metagenomics: state of the art and future[J]. Current Opinion in Microbiology, 2013, 16(3):232-239.
28.Song M, Chan A T, Fuchs C S, et al. Dietary intake of fish, ω‐3 and ω‐6 fatty acids and risk of colorectal cancer: A prospective study in U.S. men and women[J]. International Journal of Cancer, 2014 ,135(10):2413-23.
29.Tanaka T, Oyama T, Sugie S. Dietary Tricin Suppresses Inflammation-Related Colon Carcinogenesis in Mice[J]. Nutr Sci Vitaminol (Tokyo),2019;65(增刊):S100-S103.
30.Sears C , Garrett W . Microbes, Microbiota, and Colon Cancer[J]. Cell Host & Microbe, 2014, 15(3):317-328.
31. Zhang J, Guo S, Li J,et.al. Effects of high-fat diet-induced adipokines and cytokines on colorectal cancer developmenrtr[J]. FEBS Open Bio. 2019.10.30.
32.Baena R, Salinas P. Diet and colorectal cancer[J]. Maturitas. 2015,80(3):258-64.
33.Zhao Z, Yin Z, Zhao Q. Red and processed meat consumption and gastric cancer risk: a systematic review and meta-analysis[J]. Oncotarget, 2017, 8(18):30563-30575.
34.Iida N, Dzutsev A, Stewart CA,et.al.Commensal Bacteria Control Cancer Response to Therapy by Modulating the Tumor Microenvironment[J]. Science, 2013, 342(6161):967-970.
35.Gui Q F , Lu H F , Zhang C X , et al. Well-balanced commensal microbiota contributes to anti-cancer response in a lung cancer mouse model[J]. Genetics and molecular research: GMR, 2015, 14(2):5642-5651.
36. Chen ZF, Ai LY, Wang JL, et.al. Probiotics Clostridium butyricum andBacillus subtilis ameliorate intestinal tumorigenesisl[J]. Future Microbiol .2015,10(9):1433-45.
37.Lenoir M , Del Carmen S , Cortes-Perez N G , et al. Lactobacillus caseiBL23 regulates Tregand Th17 T-cell populations and reduces DMH-associated colorectal cancer[J]. Journal of Gastroenterology, 2016, 51(9):862-873.
38.Khoruts A, Rank KM, Newman KM, et.al. Inflammatory Bowel Disease Affects the Outcome of Fecal Microbiota Transplantation for Recurrent Clostridium difficile Infection[J]. Clin Gastroenterol Hepatol. 2016,14(10):1433-8.
39.Lev-Sagie A, Goldman-Wohl D, Cohen Y,et.al. Vaginal microbiometransplantation in women with intractable bacterial vaginosis[J]. Nat Med. 2019,25(10):1500-1504.
40.Sbahi H , Di Palma J A . Faecal microbiota transplantation: applications and limitations in treating gastrointestinal disorders[J]. BMJ Open Gastroenterology, 2016, 3(1):e000087.

